世界の科学者がハイパフォーマンス コンピューティング システムと NVIDIA Clara Parabricks を使ってゲノミクス データを読み解く
科学者やヘルスケアの研究者は、ウェットラボで観察できるサンプル数や細胞をのぞき込む顕微鏡の質に制約を受けていたものですが、今や強力な計算ツールを活用し、留まるところを知らぬ生物学データによる発見から知見を引き出しています。
バイオのデジタル革命を支えているのは、ハイパフォーマンス コンピューティング システムと、ドメイン特化型のソフトウェア フレームワークの組み合わせです。
最も強力なシステムの最新の TOP500 ランキングが発表され、ヘルスケアに重点を置いた NVIDIA の Cambridge-1 とバイオテクノロジー企業 Recursion 社の BioHive-1 がランクインしています。この 2 つのスーパーコンピューターは、NVIDIA DGX SuperPOD リファレンス アーキテクチャをベースにしています。
そして、世界中の医療研究機関、製薬会社、バイオテクのスタートアップは、ゲノミクス ライブラリとリファレンス アプリケーションのスイートである NVIDIA Clara Parabricks を使用して、次世代の配列解析ワークフローを強化しています。
上海を本拠地とする Mingma Biotechnology 社は、6月、精密医療の研究を推進するために 中国の研究所として初めて Clara Parabricks Pipelines を導入しました。これは、タイと日本で今年開始された大規模なゲノミクスの取り組みに続くものです。また、遺伝子治療のスタートアップである Greffex 社も Parabricks Pipelines を最近採用し、ユニバーサル インフルエンザ ワクチンの開発プロジェクトを加速させています。
集団研究のためのゲノムに関する知見を特定する
Parabricks Pipelines によって、DNA および RNA ベースのプロジェクトは NVIDIA GPU で最大 50 倍高速化されるため、科学者たちは、医療機器から日々生成される何百テラバイトものデータから、有用な情報を多数取り出すことができます。集団研究にはきわめて多数のゲノム解析が必要ですが、このような研究に取り組む公衆衛生の研究機関や研究所では特に、この高速化が大きな原動力となります。
Mingma Biotechnology 社は Parabricks Pipelines と NVIDIA T4 Tensor コア GPU を採用し、配列解析とマルチオミクス データ分析の作業を迅速に進めています。同社では、医療機関、製薬会社、および研究者たちに、病気の研究や創薬に有用なゲノムに関する知見を提供しています。
National Biobank of Thailand では、NVIDIA DGX A100 システムを利用し、Genomics Thailand という取り組みを強化しています。この取り組みは、国内共通のヘルスケア サービスとして、ゲノム医療を導入するものです。この研究機関では、5 万人のタイ人ボランティアのゲノム配列解析データ全体を利用して遺伝子変異を分析するために Parabricks Pipelines を使用しています。
DGX システムと Parabricks Pipelines を組み合わせることで、プロジェクト全体のゲノム データの処理時間が 4 カ月短縮されました。この研究から得られる知見は、タイ国民に特有の遺伝子変異をより適切に分析するのに役立つことが期待されます。
日本では、国立大学法人東京大学医科学研究所のヒトゲノム解析センターで、最新型のヒトゲノム解析用スーパーコンピューターである SHIROKANE にDGX A100を搭載し、Parabricks Pipelines を実行しています。がん研究に貢献するヒトゲノム情報の効率的な解析と個別化医療の実現を目的に運用されています。
クリニカル シーケンシングや創薬に力を発揮
Parabricks Pipelines の一連の遺伝学的ツールは、各研究室固有のニーズに合わせて構成できます。研究者たちは、デスクトップ ワークステーションから GPU アクセラレーテッド クラウドや一部の世界最速のスーパーコンピューターまで、Parabricks Pipelines の幅広いワークロードを NVIDIA GPU システムで実行しています。
米国ヒューストンを本拠地とする Greffex 社は、NVIDIA RTX データサイエンス ワークステーションを利用開始してから数週間のうちに、Parabricks Pipelines と NVIDIA Clara Discovery の利用を開始し、ユニバーサル インフルエンザ ワクチンの開発作業を推進しています。
このスタートアップ企業は、ゲノム配列解析、分子動力学ツール、ウェットラボでの研究を組み合わせ、時間の経過に伴ってインフルエンザ株がどのように進化するのか、これらの変異株がワクチンの有効性にどのような影響を与えるのかを研究しています。
Greffex 社では、インフルエンザ ウイルスの変異を観察するために、世界中から膨大な数のインフルエンザ ゲノムを収集し、NVIDIA RTX 8000 GPU で配列アラインメントを大規模に実行して、ウイルスの遺伝情報が変化している場所を特定しています。GPU でゲノムのワークロードを実行すると、1 サンプルあたり最大 13 時間の節約になり、アラインメント結果を微調整するために別のパラメーターでサンプルを再実行することもできるようになります。
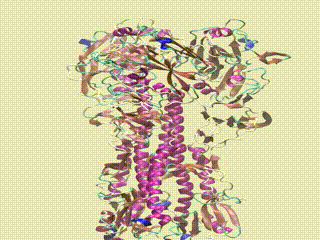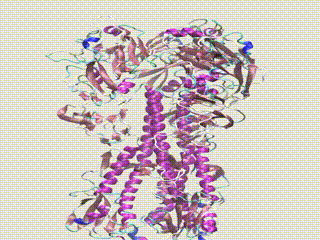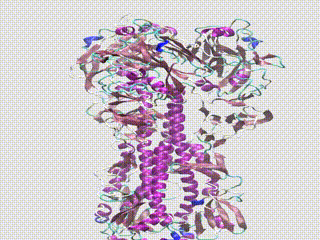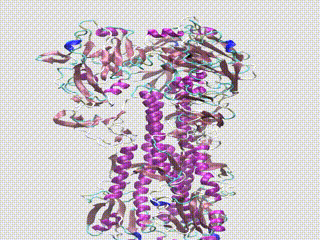
Greffex 社の科学者たちは、インフルエンザ ウイルスの表面に存在するタンパク質のヘマグルチニンに対して
数値計算による分子動力学シミュレーションを実行し、自然環境でどのような動きをするか観察しています。
Greffex 社の科学者たちは、遺伝的変異体が特定されると、このような遺伝的変異によってインフルエンザ ウイルスの物理的形状がどのように変化したのかを、分子動力学を活用して可視化します。彼らは、インフルエンザ ウイルスが、ワクチンによって誘導された抗体と結合しない形状に少しずつ変化していく可能性のある、分岐変異株を監視しています。
Greffex 社の生物情報科学者であるダニエル プレストン (Daniel Preston) 氏は、次のように述べています。「インフルエンザ ウイルスの現在の株だけでなく、その他多くの株と結合するワクチンのタンパク質構造を最適化するプロセスには、非常に長い時間と膨大なコストがかかります。計算手法があれば、実際の研究室でテストをする前に、何が機能する可能性が高いのか、見当を付けることができます。例えるなら、ハンマーを使っていたのが外科用のメスになったくらいの進化です。」
NVIDIA Clara Parabricks について
NVIDIA Clara Parabricks は、Broad Institute の業界標準となっている Genome Analysis Toolkit や Google 社の DeepVariant バリアント コーラーなど、普及しているツールを GPU で高速化します。Parabricks を NVIDIA A100 Tensor コア GPU で実行すると、全ヒトゲノムの二次解析で生殖細胞系列の遺伝子変異検出は 23 分で完了します。これは、CPU システムで実行すると 20 時間以上かかる処理です。
Clara Parabricks Pipelines では、DNA の配列読み取りをはじめ、アラインメント、ソート、フィルター、生殖細胞変異検出、体細胞変異検出、RNA ベースのアプリケーションのサポートが可能です。生殖細胞変異は個々の家系を通じて継承されるのに対して、体細胞変異は人が生涯を送る間にヒト細胞内で発生し、がんを引き起こす可能性があります。
来月リリースされる Parabricks Pipelines バージョン 3.6 では、体細胞変異検出のツールをさらに拡充し、研究者に精密腫瘍学向けの知見を提供するとともに、生殖細胞系の de novo 変異検出にも対応し、自閉症などの複雑な疾患の研究にも情報を提供します。
生殖細胞系の de novo 変異検出パイプラインは、米国ワシントン大学医学部の研究者の協力により開発されました。この技術により、ゲノム データの解析と、家系または親子トリオ内の新しい変異の特定にかかる処理時間を 1 時間以内に短縮できます。
NGC または AWS Marketplace から、ゲノム解析を加速させる NVIDIA Clara Parabricks Pipelines をぜひご利用ください。
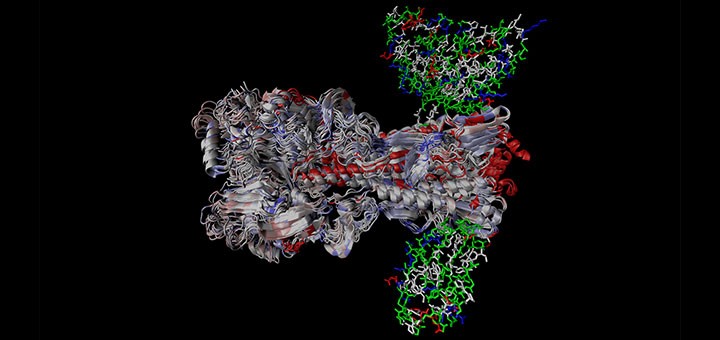
ブログトップのメイン画像は、102 年の歳月を経た 17 H1 ヘマグルチニン タンパク質の構造アラインメントと結合した抗体のシミュレーションを示しています。タンパク質内の突然変異株の種類に対応して色分けされています。画像提供:Greffex 社、RCSB Protein Data Bank のモデルを使用。

 Chrome
Chrome Firefox
Firefox Opera
Opera Safari
Safari IE
IE