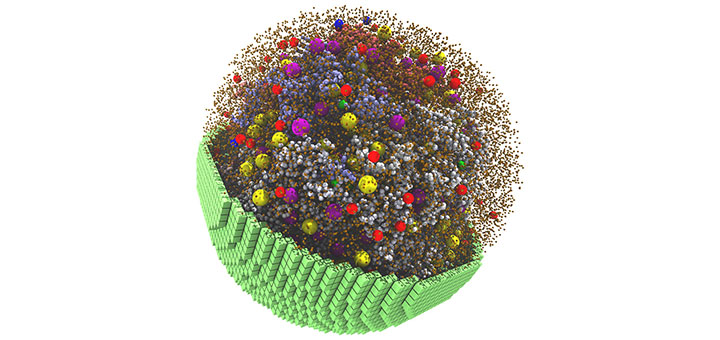
イリノイ大学アーバナ シャンペーン校の研究者たちが開発したGPU アクセラレーテッド ソフトウェアにより、生細胞のように代謝し成長する 20 億個の原子を持つ細胞のシミュレーションを実現
すべての生細胞は、エネルギー生産、タンパク質合成、遺伝子転写などに関わる何千もの構成要素が含まれています。
イリノイ大学アーバナ シャンペーン校の科学者たちは、こうした物理的特性および化学的特性を粒子スケールで再現する 3D シミュレーションを構築しました。生細胞の挙動を模倣する、完全に動的なモデルを作成するものです。
学術誌 『Cell』 に掲載されたこのプロジェクトでは、細胞の生存、機能、複製に不可欠な遺伝子を含む最小生細胞をシミュレートするものです。このモデルでは NVIDIA の GPU を使用し、20 分間の細胞周期にわたって 7,000 の遺伝子情報伝達のシミュレーションを行います。これにより、科学者たちにとってこれまでで最長かつ最も複雑な細胞シミュレーションと考えられるものが実現します。
最小細胞は、自然に存在する細胞より単純であるため、デジタルでの再現がより容易です。
同大学の生体細胞物理学センターで化学教授および共同センター長を務めるザイダ ルセイ=シュルテン (Zaida Luthey-Schulten) 氏は次のように述べています。「最小細胞であっても、20 億個の原子が必要です。GPU がなければ、このような 3D モデルを現実的な人間の時間スケールで作成することはできません。」
さらなるテストと改良が完了すれば、全細胞モデルを用いて、実世界の細胞の状態もしくはゲノムに対する変化がその機能にどのように影響を及ぼすかを科学者たちが予測できるようになります。しかしこの段階であっても、最小細胞のシミュレーションにより、科学者たちは生細胞の基盤を形成する物理的プロセスおよび化学的プロセスについて、洞察を得ることができます。
「わかったことは、シミュレーションされた細胞から、基本的な振る舞いが現れるということです。それをプログラムとして組み込んだからではなく、私たちのモデルにおける動態パラメーターと脂質メカニズムが適切だったからです。」と、ルセイ=シュルテン氏は述べています。
ルセイ=シュルテン氏が共同開発し、3D 最小細胞のシミュレーションに使用される、GPUアクセラレーテッド ソフトウェアである Lattice Microbes は、NVIDIA NGC ソフトウェア ハブから利用可能です。
最大のリアリズムを備えた最小細胞
生細胞モデルを構築するため、イリノイ大学の研究者たちは最も単純な生細胞の 1 つである、マイコプラズマと呼ばれる寄生細菌のシミュレーションを行いました。このモデルは、カリフォルニア州ラホヤにある J. クレイグ ベンダー研究所の科学者たちが合成したマイコプラズマ細胞の縮小版で、生存に必要な遺伝子はわずか500個弱でした。
比較すると、単一の大腸菌細胞には約 5,000 個の遺伝子があります。人間の細胞 1 つでは 2 万個以上です。
次いでルセイ=シュルテン氏のチームは、アミノ酸、ヌクレオチド、脂質、小分子代謝物など、マイコプラズマの内部構造が持つ既知の性質を使用して、DNA、RNA、タンパク質、細胞膜を備えたモデルを構築しました。
「既知のものすべてを再現するのに十分な反応を得ることができました。」と、彼女は述べています。
研究者たちは NVIDIA Tensor コア GPU 上で Lattice Microbes ソフトウェアを使用し、細胞が大幅に拡大したりDNAを複製したりするライフ サイクルを 20 分間、 3D シミュレーションを実行しました。その後、細胞の DNA の大幅な拡張もしくは複製が始まります。モデルにより、細胞は細胞膜を超えて分子を輸送することに、そのエネルギーの大部分を費やしていることが示されました。これは寄生細胞としてのプロファイルに合致するものです。
大学院生であり論文の筆頭著者であるザーネ ソーンバーグ (Zane Thornburg) 氏は次のように述べています。「こうした計算を連続的に、もしくは全原子レベルで行うとすれば、数年かかるでしょう。しかし、計算がすべて独立したプロセスであるため、GPUによるコードの並列処理を採用することができました。」
ソーンバーグ氏は、3D で成長と細胞分裂のシミュレーションを行うプロジェクトに取り組んでおり、こちらもGPUによって高速化しています。チームは最近、作業をさらに加速するため NVIDIA DGX システムと RTX A500 GPU を導入し、A5000 GPU を使用することで、前世代の NVIDIA GPU を搭載した開発ワークステーションと比較して、ベンチマーク シミュレーション時間が 40% 高速化されることがわかりました。
3 月 21 日~ 24 日にオンラインで開催される NVIDIA GTC に無料登録し、NVIDIA の GPU を使用した科学の飛躍的進歩を加速する研究者たちについての詳細をご確認ください。
※本ブログのトップ画像は、20 分間の 3D 空間シミュレーションからの1コマです。黄色と紫色のリボソーム、赤色と青色のデグラドソームが示されており、より小さな球体は DNA ポリマーとタンパク質を表すものです。