オークリッジの研究者が、Summit スーパーコンピューターを使って探索時間を数年から数時間に短縮。
在宅で、ときにパジャマで仕事をしながら、エイダ セドヴァ (Ada Sedova) 氏は世界で最も強力なスーパーコンピューターにアクセスして、新型コロナウイルスによる COVID-19 感染を止める可能性のある小さな分子を探します。
米国オークリッジ国立研究所 (ORNL) で生物物理学を研究するセドヴァ氏は次のように述べています。「これまでにないほど多くのことを成し遂げています。パンデミックをめぐる不安はありますが、私の個人的な時間の多くをこの取り組みにささげています」
同氏の努力は 10 億ドル単位の収入をもたらすほどのものです。具体的には、わずか 24 時間で 20 億回の分子テストを実行しました。
セドヴァ氏は、数十個の原子よりも小さな有機分子であるリガンドを探しています。適切なリガンドは新型コロナウイルスのタンパク質に付着して、健康な細胞へのウイルス感染を防ぐと考えられます。
問題は、調べるべきリガンドとタンパク質があまりに多いことと、どちらも原子の力の変化に合わせて形状が変わり続けることです。これは、何十億もの膨大な候補の化合物の山から、1 本の小さな針を探すようなものです。
候補の 1 つ 1 つを専門家がウェット ラボで試すとしたら、何十年もかかるでしょう。そのすべてを、ORNL のスーパーコンピューターである Summit に搭載の 9,216 基 の CPUでシミュレートしたとしても、4 年はかかるでしょう。そこでセドヴァ氏と同僚は、この取り組みを加速するために、Summit の 27,648 基の NVIDIA GPU に頼ることにしました。
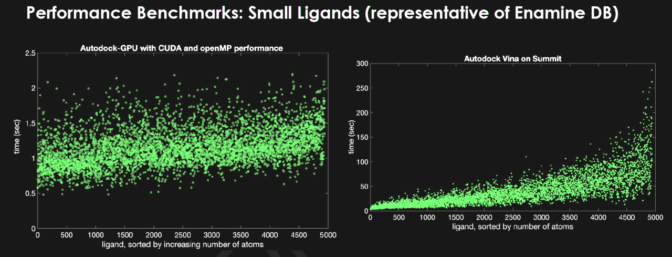
同氏らは、スクリプス研究所がダルムシュタット工科大学との共同研究で開発された AutoDock (タンパク質とリガンドがどのように結合するかをシミュレートするオープンソース プログラム) の OpenCL バージョンを使い始めました。GPU 上で OpenCL バージョンを使うことで、CPU と比べて処理速度が 50 倍に向上しました。
CUDA はずばり要点を突く
NVIDIA とスクリプス研究所の協力を得て、チームはコードを CUDA に移植して Summit で実行できるようにしました。これには、処理速度がさらに 2.8 倍向上するというメリットもありました。また別の研究者、Jubilee Development のアーロン シャインバーグ (Aaron Scheinberg) 氏が、OpenMP を使って GPU へのデータ供給を高速化する方法を発見し、処理はさらに 3 倍速くなりました。
セドヴァ氏は別のテストにおいて、1 つのタンパク質に対し、14 億の化合物から成るデータセットを、わずか 12 時間で高い精度でスクリーニングできる可能性を示唆する結果を示しました。これは、プログラムが CPU で実行される場合に比べて 33 倍以上の高速化です。
セドヴァ氏は次のように述べています。「GPU を Summit の規模やアーキテクチャと組み合わせることで、以前よりもさらに数十億の化合物をドッキングできるようになります」
チームメンバーの 1 人である生物物理学者のジョシュ ファーマース (Josh Vermaas) 氏は、AutoDock の CUDA への移植を支援した NVIDIA の スコット ル グラン (Scott Le Grand) に感謝を述べました。プロジェクトの始まりについて語るブログの中で、ファーマース氏は次のように述べています。「OpenCL のみに対応していたコードのパフォーマンスを改善する上で、彼は並外れた支援をしてくれました」
24 時間で 20 億の化合物をシミュレーション
セドヴァ氏は現在、さらなる改善を加えれば、チームは 24 時間で 20 億もの化合物を調査できるのではないかと考えています。これを実現すれば、高解像度で行われるこの規模のシミュレーションとしては初となるでしょう。
そのマイルストーンを目指す上で、研究者はまだいくつかの課題に直面しています。
タンパク質-リガンド ドッキングの標準的なワークフローには、ファイルベースの遅いプロセスが使われています。ノート PC で数百の化合物をテストする程度ならばこれでもかまいませんが、ファイルが何十万個もあるような規模の場合、世界最大のスーパーコンピューターでも滞るでしょう。
これは、科学の加速を支援したいと考えるオープンソース開発者へ行動を呼びかけるものです。
セドヴァ氏のチームは先頭に立って、Summit 上で行われる大量のジョブを安全に起動できる新しいワークフローを組み立てています。同氏は Summit システムの I/O 専門家に相談して、すべてのリガンドのデータを保持するデータベースを立ち上げようとしています。
次のステップは、Summit の 4,608 ノードのうちの 108 ノードで、約 100 万の化合物を試す実験を始めることです。「これがうまくいったら、Summit の全ノードを使って 14 億の化合物を試す大規模な実験を開始します」とセドヴァ氏は述べています。
有望な分子に向けて探索を絞り込む
もしチームが成功した場合、チームは米国テネシー州メンフィスにいる研究者に、最も有望な化合物約 9,000 件のリストを送り、ウェット ラボで本物のウイルスを使ったテストを実施してもらう予定です。これは干し草の山の中の針ではなく、シャベル 1 杯分の干し草の中の針と言えます。
このプロジェクトは、今年の 1 月、ORNL のトップ研究者であるジェレミー C スミス (Jeremy C. Smith) 氏が、新型コロナウイルスと戦うための創薬研究に初めて Summit スーパーコンピューターを使った成果を示したときに始まりました。この取り組みはまだ初期段階にあります。
将来に向けて、セドヴァ氏には、タンパク質-リガンド結合の分野を、ハイパフォーマンス コンピューティングで使われている典型的な手法と結びつける別の方法のアイデアがあります。そしてもちろん、それを追究するエネルギーにもあふれています。
新型コロナウイルスとの戦いに GPU を活用するさまざまな取り組みについて、NVIDIA の COVID-19 ページをご覧ください。

 Chrome
Chrome Firefox
Firefox Opera
Opera Safari
Safari IE
IE